El Análisis de componentes principales es una técnica muy útil cuando tenemos datos que contienen múltiples variables, y que queremos reducir con el fin de quedarnos solo con lo esencial.

Contenido del post
Introducción
El Análisis de componentes principales, o PCA por sus siglas en inglés, es una técnica muy útil cuando tenemos que trabajar con datos que tienen muchas variables o características. Por ejemplo, imagina que estás estudiando un ecosistema y tienes datos de la temperatura, la humedad, la vegetación, el viento, el riesgo de incendios, etc. ¿Cómo puedes visualizar toda esa información en un solo gráfico? ¿Cómo puedes saber qué variables son las más importantes o las que más influyen en el comportamiento del ecosistema? Aquí es donde entra el PCA.
El PCA es una forma de reducir la dimensionalidad de los datos, es decir, de simplificarlos para quedarnos solo con lo esencial. Lo que hace el PCA es encontrar unas nuevas variables, llamadas componentes principales, que son combinaciones lineales de las variables originales, que tienen la propiedad de capturar la mayor parte de la variación o la información de los datos. Así, podemos representar los datos en un espacio de menor dimensión, por ejemplo, en un plano bidimensional o en un espacio tridimensional. Esto nos puede dar pistas sobre qué variables son las que determinan el fenómeno del sistema que estamos estudiando. Por ejemplo, si la primera componente principal está muy relacionada con la temperatura y la humedad, podemos inferir que estas variables son las que más afectan al ecosistema.
En resumen, el PCA es una técnica muy poderosa y versátil que nos permite simplificar y entender mejor nuestros datos. Si quieres aprender más sobre cómo aplicar el PCA a tus datos, te invito a seguir leyendo este blog, te explicaremos cómo hacer el PCA paso a paso en R y cómo interpretar los resultados.

Código en R
Para aprender a hacer un PCA en R usaremos un conjunto de datos llamado bioenv, obtenido del blog Principal Components Analysis.
Como primera medida a tomar, conoceremos las paqueterías de R que vamos a utilizar en este ejercicio.
library(readxl)
library(GGally)
library(factoextra)
library(corrplot)Esta base de datos, es un conjunto de variables observadas en lugares del fondo marino, en un estudio de biología marina: el primero es un conjunto de variables biológicas, el recuento de cinco grupos de especies, y el segundo es un conjunto de cuatro variables ambientales. Los grupos de especies se abrevian como “a” hasta “e”. Las variables ambientales son “contaminación”, un índice compuesto de contaminación que combina mediciones de concentraciones de metales pesados e hidrocarburos; profundidad, la profundidad en metros del fondo marino donde se tomó la muestra; “temperatura”, la temperatura del agua en el punto de muestreo; y ‘sedimento’, una clasificación del sustrato de la muestra en una de tres categorías de sedimentos.

Para el ejercicio que realizaremos únicamente analizaremos las variables del recuendo de los cinco grupos de especies. Así, comenzaremos leyendo nuestra base de datos desde nuestro repositorio en GITHUB, y filtraremos la información que necesitemos.
## Esta es una base de datos que se encuentra en línea, por ello,
## debemos guardar los datos en una variable
bioenv_data <- read.csv('https://raw.githubusercontent.com/RBiologos/Docs/main/bioenv.csv', sep = ';')
## Veamos un poco de nuestros datos
head(bioenv_data)## Site a b c d e Pollution Depth Temperature Sediment
## 1 s1 0 2 9 14 2 4.8 72 3.5 S
## 2 s2 26 4 13 11 0 2.8 75 2.5 C
## 3 s3 0 10 9 8 0 5.4 59 2.7 C
## 4 s4 0 0 15 3 0 8.2 64 2.9 S
## 5 s5 13 5 3 10 7 3.9 61 3.1 C
## 6 s6 31 21 13 16 5 2.6 94 3.5 G## Eliminamos variables que no necesitamos
bioenv <- bioenv_data[, 2:6]Ahora, hagámos una breve incursión por la naturaleza de nuestros datos y su comportamiento. Para ello, analizaremos su media y varianza, con el fin de encontrar posibles problemas para el análisis de nuestro PCA.
## Análisis de media
apply(X = bioenv, MARGIN = 2, FUN = mean)## a b c d e
## 13.466667 8.733333 8.400000 10.900000 2.966667## Análisis de varianza
apply(X = bioenv, MARGIN = 2, FUN = var)## a b c d e
## 157.63678 83.44368 73.62759 44.43793 15.68851Como observamos, el promedio de los datos muestra que no hay estandarización entre las variables, mientras que variables como Depth quien posee valores muy altos en comparación con las otra variables.Además podemos observar que en términos de la varianza, la variable Depth sigue teniendo el mismo comportamiento. Si no se estandarizan las variables para que tengan media cero y desviación estándar 1 antes de realizar el estudio PCA, la variable Depth dominará la mayoría de las componentes principales.
Análisis de correlación
En el primer paso para el entendimiento de nuestros datos, haremos una correlación, la cual suele usarse para analizar la correlación de las variables que tenemos previamente al análisis de PCA. Se puede calcular usando la función
## Ejecutamos el código para la correlación de las variables
corr <- ggpairs(bioenv)
## Graficamos dicho resultado
corr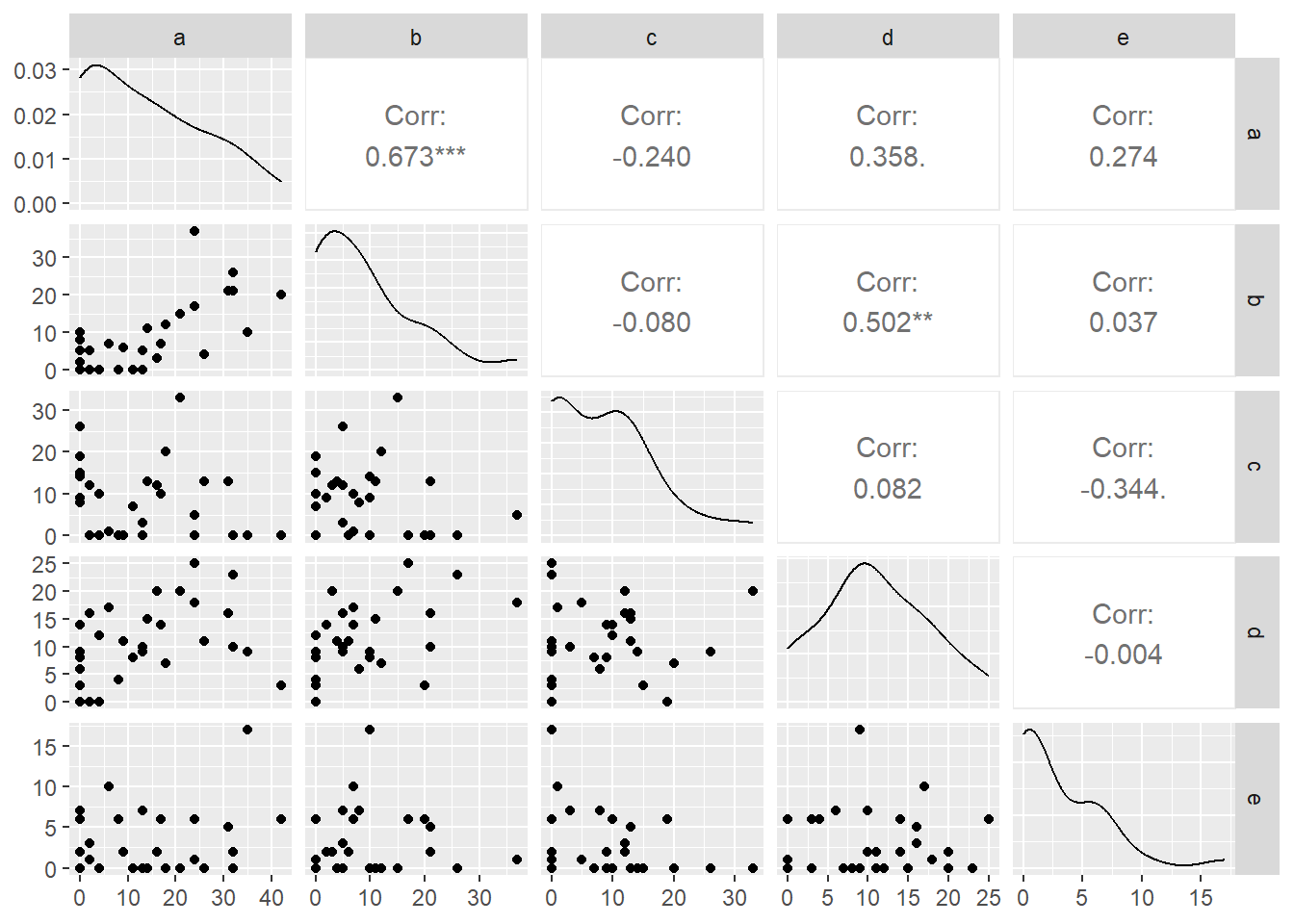
El resultado de la matriz de correlación se puede interpretar de la siguiente manera:
- Cuanto mayor sea el valor, más correlacionadas positivamente estarán las dos variables.
- Cuanto más cerca esté el valor de -1, más negativamente estarán correlacionados.
Así podemos decir que las variables tienen una correlación alta, siendo la variable a y b poseen los mayores valores. Cabe resaltar que la variable c tiene los valores más bajos de correlación en comparación con las otras variables.
## Realizamos nuestro análisis PCA
pca <- prcomp(bioenv, center=TRUE, retx=TRUE)
## Visualizamos el resumen de los resultados
summary(pca)## Importance of components:
## PC1 PC2 PC3 PC4 PC5
## Standard deviation 14.8653 8.8149 6.2193 5.03477 3.48231
## Proportion of Variance 0.5895 0.2073 0.1032 0.06763 0.03235
## Cumulative Proportion 0.5895 0.7968 0.9000 0.96765 1.00000Vemos que aproximadamente el 58.95 % de la variación en los datos es capturada por el primer componente (PC1) y aproximadamente el 90.00 % por los tres primeros componentes (PC3).
Comparemos los valores devueltos por el PCA con los que obtendríamos si realizáramos un análisis propio de la matriz de covarianza que corresponde a nuestros datos. De esta manera, procederemos a ver nuestros resultados
pca## Standard deviations (1, .., p=5):
## [1] 14.865306 8.814912 6.219250 5.034774 3.482308
##
## Rotation (n x k) = (5 x 5):
## PC1 PC2 PC3 PC4 PC5
## a 0.81064462 0.07052882 -0.53108427 0.18442140 -0.14771336
## b 0.51264394 -0.27799671 0.47711910 -0.63418946 0.17342177
## c -0.16235135 -0.88665551 -0.40897655 -0.01149647 0.14173943
## d 0.22207108 -0.31665237 0.56250980 0.72941223 -0.04422938
## e 0.06616623 0.17696554 -0.08141111 0.17781482 0.96231977Visualización de los componentes principales
El análisis previo de la matriz de carga proporcionó una buena comprensión de la relación entre cada uno de los dos primeros componentes principales y los atributos de los datos. Sin embargo, puede que no resulte atractivo visualmente. Para entender mejor estos resultados hay un par de estrategias de visualización estándar que pueden ayudar al investigador a obtener información sobre los datos.

Scree Plot
Una de estas herramientas se utiliza para visualizar la importancia de cada componente principal y se puede utilizar para determinar la cantidad de componentes principales que se deben mantener en un análisis de componentes principales (PCA). El Scree Plot se puede generar usando la función
fviz_eig(pca, addlabels = TRUE)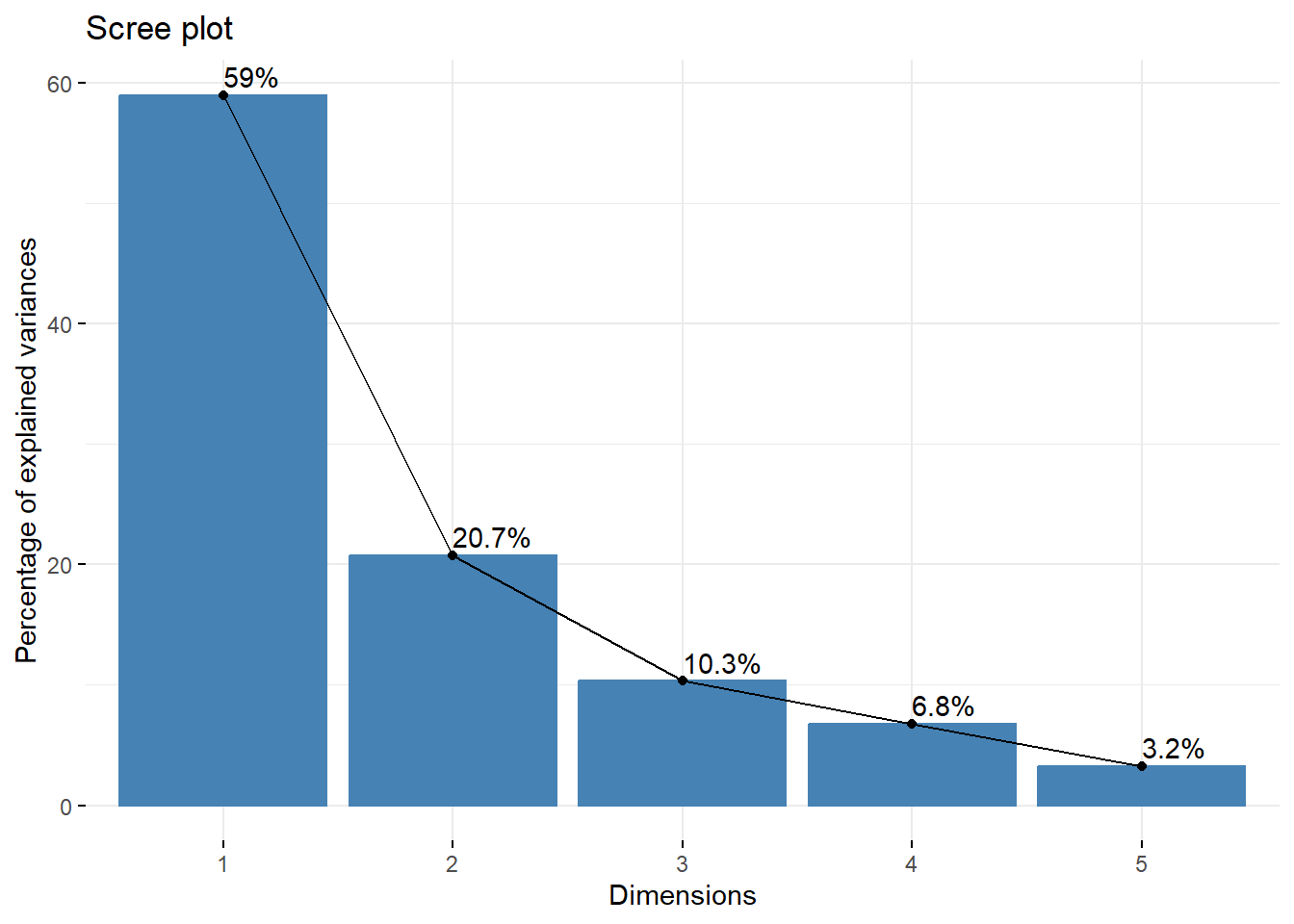
Este gráfico muestra los valores propios en una curva descendente, de mayor a menor. Los dos primeros componentes pueden considerarse los más significativos ya que contienen casi el 90 % de la información total de los datos.
Biplot de los atributos
De manera paralela podemos utilizar el gráfico de biplot, con el cual es posible visualizar las similitudes y diferencias entre las muestras, y además muestra el impacto de cada atributo en cada uno de los componentes principales.
# Graph of the variables
fviz_pca_var(pca, col.var = "black")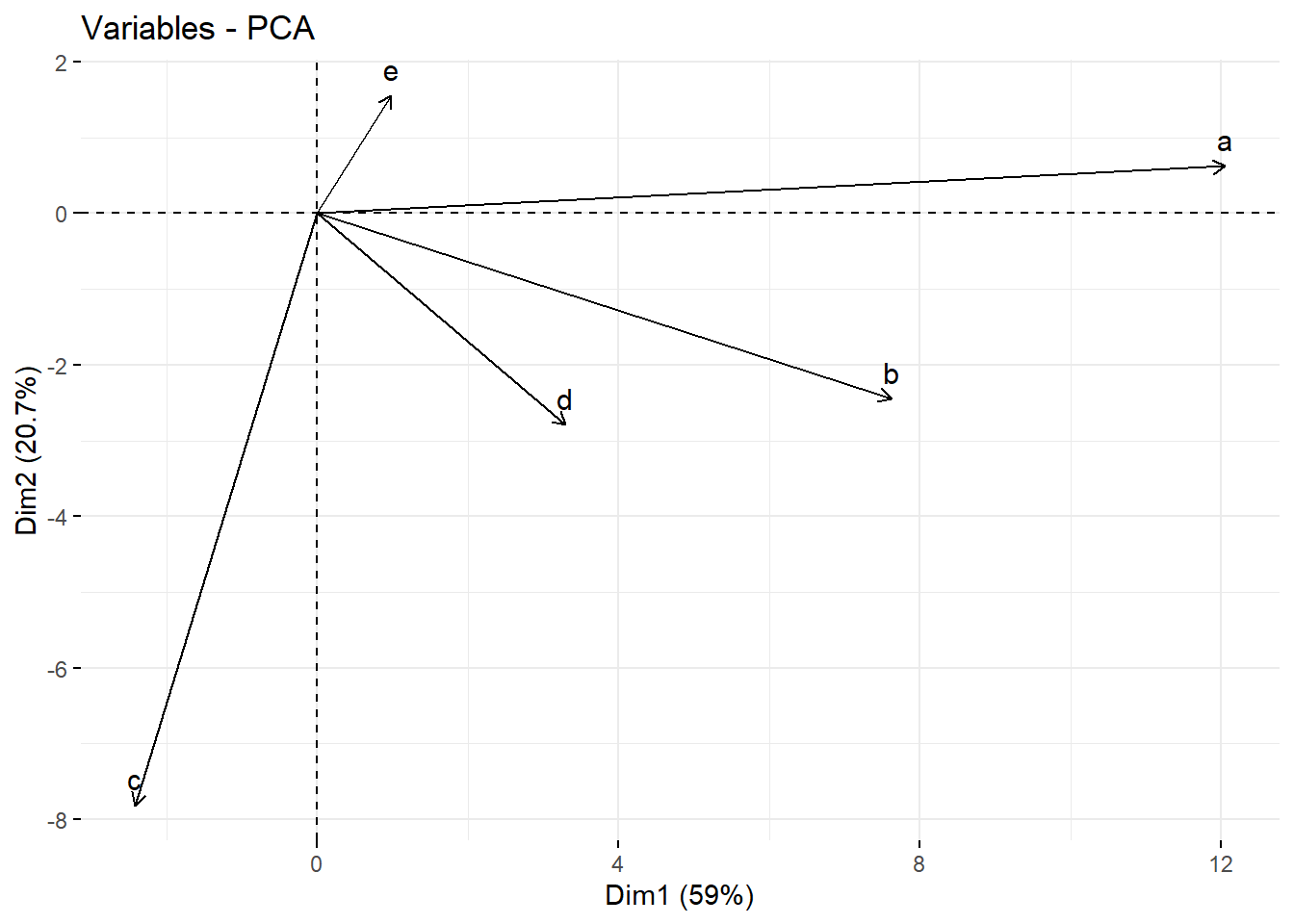
Del gráfico anterior se pueden observar tres datos principales.
En primer lugar, todas las variables que se agrupan están en cierto grado de correlación positivamente entre sí, y ese es el caso, por ejemplo, de las variables a, b y d que tienen una correlación positiva entre sí.
Entonces, cuanto mayor sea la distancia entre la variable y el origen, mejor representada estará esa variable. Según el biplot, la variable a tiene una magnitud mayor en comparación con las variables b y d y, por lo tanto, están bien representados en comparación con la variable a.
Finalmente, las variables que están correlacionadas negativamente se muestran en los lados opuestos del origen del biplot, como la variable c.
Igualmente, a partir del biplot podemos inferir que la variable a está altamente correlacionada con PC1, pero solo débilmente asociada con PC2. Por el contrario, la variable c está fuertemente correlacionada con PC2 pero sólo débilmente con PC1. También podemos aproximar las correlaciones entre las variables mismas; por ejemplo, b y d están bastante fuertemente correlacionadas, pero débilmente correlacionadas con c.

Calcular cargas factoriales
Las cargas factoriales representan la fuerza de la relación entre las variables observadas y los factores subyacentes. Cabe resaltar que un error común es interpretar las cargas factoriales como correlaciones entre las variables, para poder entender mejor la importancia y los errores más comunes puedes visitar el sitio Carga factorial decodificacion de cargas factoriales en el modelo multifactor. Ahora si, calculemos las “cargas factoriales” asociadas a la PC.
V <- pca$rotation # eigenvectors
L <- diag(pca$sdev) # diag mtx w/sqrts of eigenvalues on diag.
loadings <- V %*% L
loadings## [,1] [,2] [,3] [,4] [,5]
## a 12.0504801 0.6217053 -3.3029460 0.92852016 -0.5143835
## b 7.6206090 -2.4505164 2.9673232 -3.19300085 0.6039081
## c -2.4134024 -7.8157898 -2.5435276 -0.05788214 0.4935804
## d 3.3011545 -2.7912626 3.4983893 3.67242602 -0.1540203
## e 0.9835813 1.5599356 -0.5063161 0.89525751 3.3510942La magnitud de las cargas factoriales es en lo que desea centrarse. Por ejemplo, las especies a y b contribuyen más al primer PC, mientras que la especie c tiene la mayor influencia en el PC2.
Contribución de cada variable
El objetivo de la esta visualización es determinar cuánto está representada cada variable en un componente determinado. Esta calidad de representación se llama Cos2 y corresponde al coseno cuadrado, y se calcula utilizando la función
Un valor bajo significa que la variable no está perfectamente representada por ese componente. Por otro lado, un valor alto significa una buena representación de la variable en ese componente.
## Contribución de las variables al PC1 y PC2
fviz_cos2(pca, choice = "var", axes = 1:2)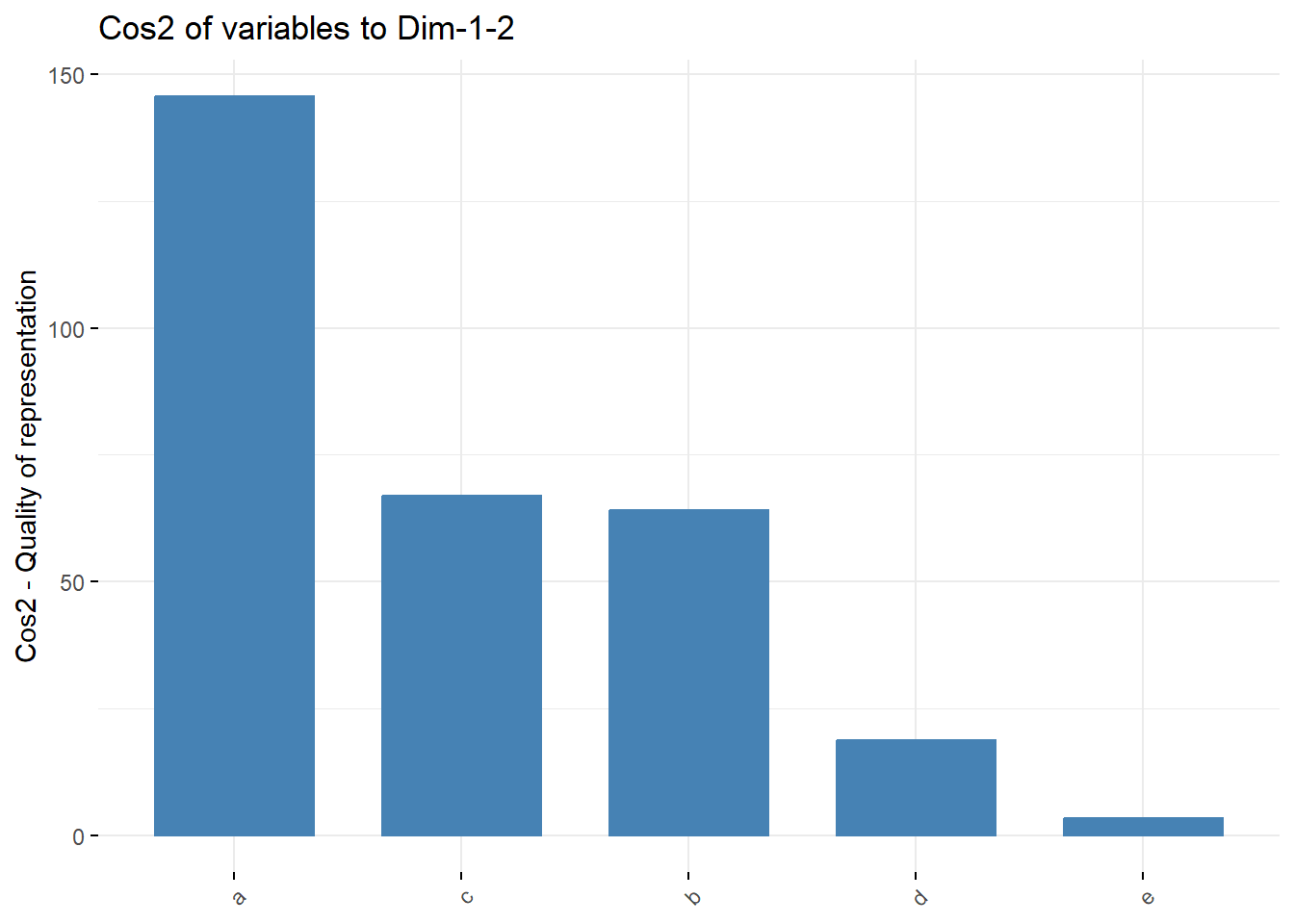
Igualmente, podemos crear un mapa de factores a partir igualmente del cos2 (coseno cuadrado, coordenadas al cuadrado), a partir de la paquetería
## Extraemos los datos del PCA para graficar
var <- get_pca_var(pca)
## Generamos el gráfico de correlación
corrplot(var$contrib, is.corr=FALSE) 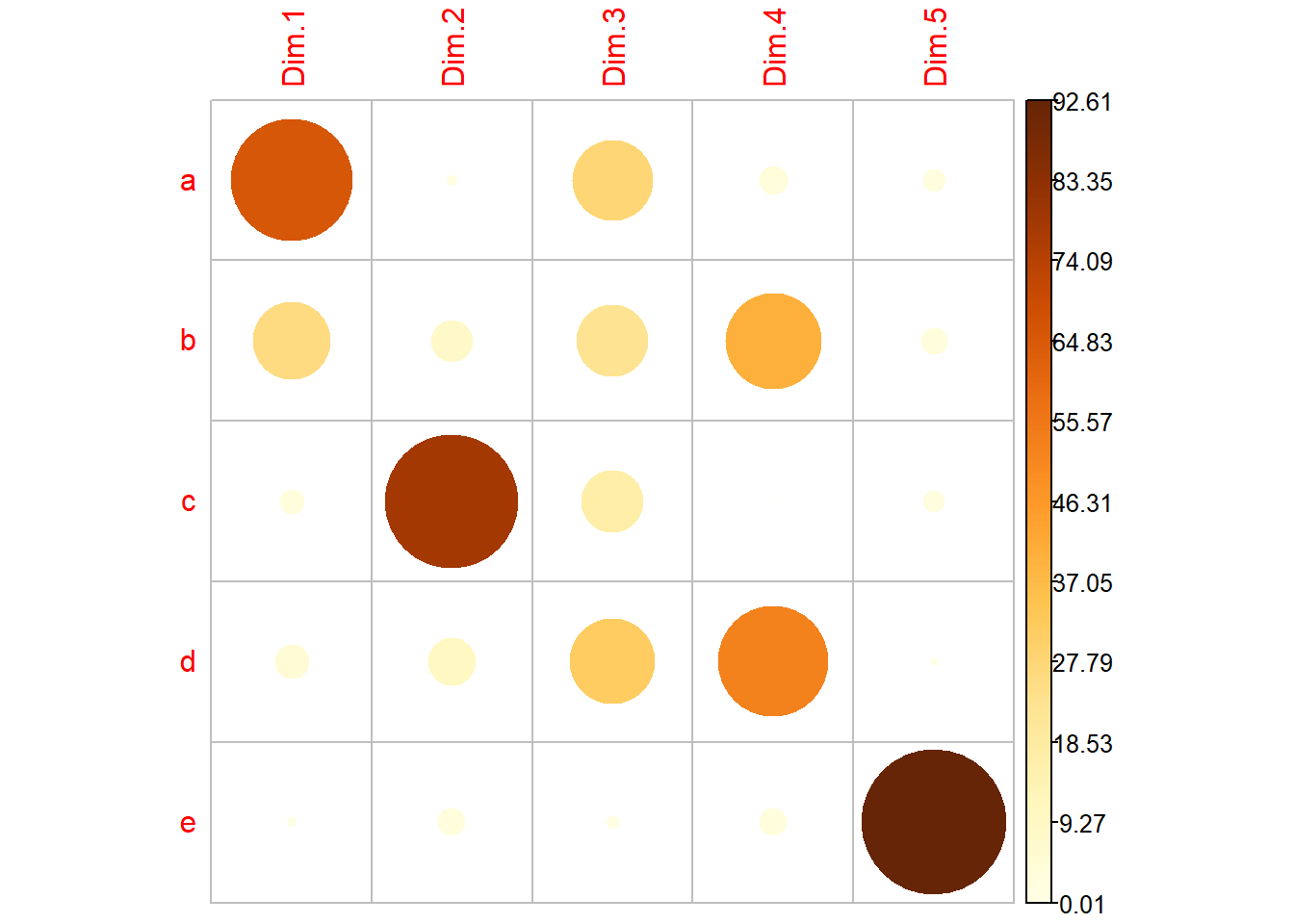
En los códigos y gráficos anteriores se calculó el valor del coseno cuadrado para cada variable con respecto a los dos primeros componentes principales.
Así, en las ilustraciones en general, las variables a, b y c son las tres variables principales con el cos2 más alto y, por lo tanto, son las que más contribuyen a PC1 y PC2. Particularmente las variables a y b contribuyen en mayor medida al PC1 y la variable c contribuye en mayor manera al PC2, en relación a los otras variables.

Biplot combinado con cos2
Los dos últimos enfoques de visualización son biplot e importancia de las variables, los cuales se pueden combinar para crear un único biplot, donde las variables con puntuaciones de cos2 similares tendrán colores similares. Esto se logra ajustando la función
fviz_pca_var(pca, col.var = "cos2",
gradient.cols = c("black", "blue", "green"),
repel = TRUE)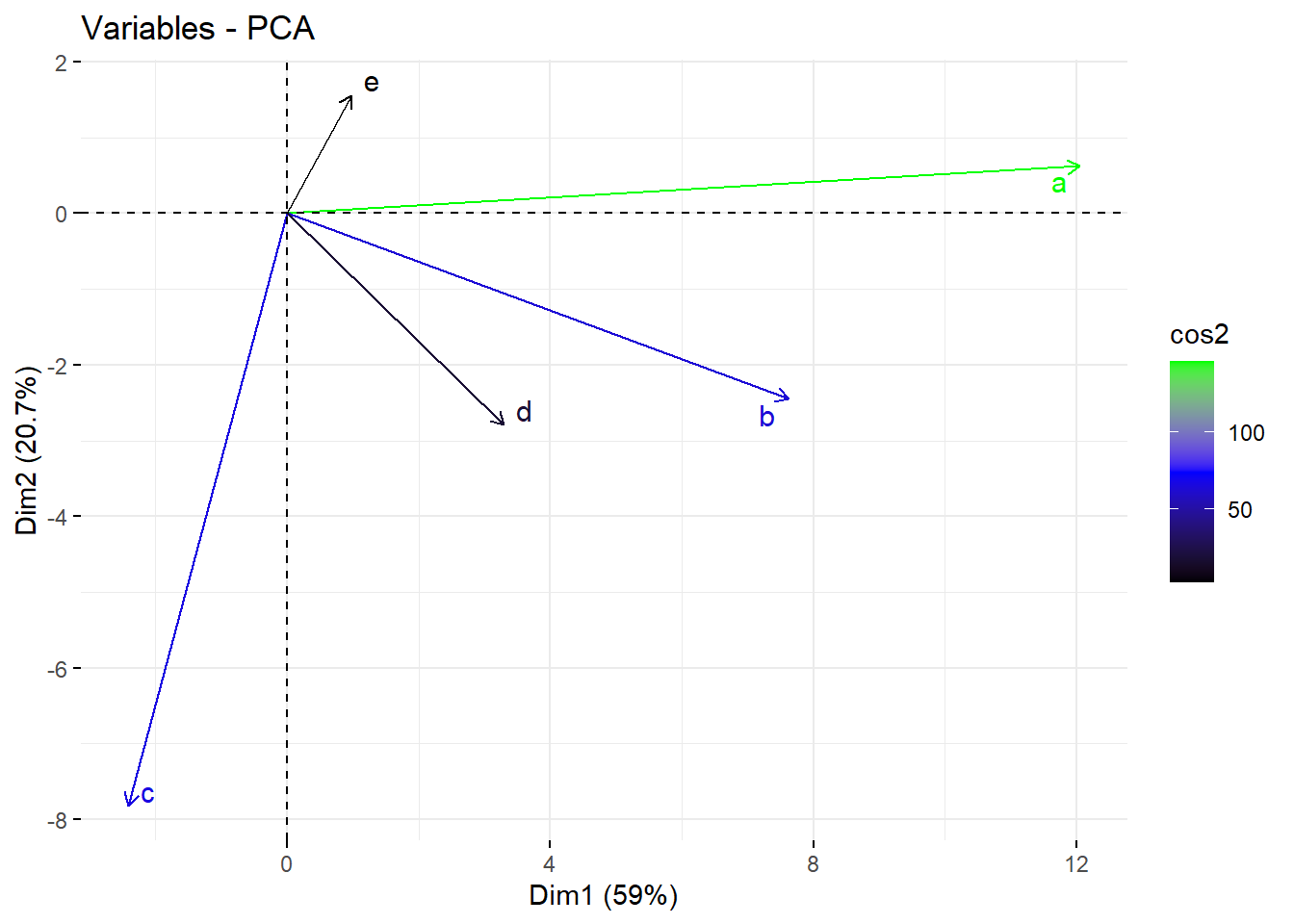
Del gráfico biplot anterior entonces podemos concluir que:
- Las variables con alto cos2 están coloreadas en verde: a.
- Ls variavles con medio cos2 tienen un color azul: b y c.
- Finalmente, las variables de bajo cos2 tienen color negro: d y e.
Resumen
Esta lectura ha cubierto una parte de qué es el Análisis de componentes principales y su importancia en el análisis de datos, a partir de datos biológicos. Así mismo, pudimos aprender algunas técnicas visuales para su entendimiento como el biplot y los resultados del cos2, con el fin de entender la relación de las distintas variables.
Como siempre, desde RBiólogos esperamos que este tipo de post nos ayuden a entrar en este fascinante mundo. Cabe resaltar que es apenas una aproximación a toda la matemática y biología que hay detras de ello. Por eso, te invitamos a que puedas profundizar más en este tipo de análisis para poder tomar las mejores decisiones en tu proyecto.
Así mismo, recordar que la estadística y matemática deben ir acompañadas por una buena discusión biológica, donde se pueda hacer ciencia de excelente calidad.

Más información
Estos análisis se han realizado utilizando el software R (v.4.3.2) y Rstudio (v. 2023.12.0)
Recuerda que todos nuestros códigos están almacenados en GitHub


